選自New Relic
作者:Jason Snell
參與:Panda、劉曉坤
時間序列數據(Time Series Data)是按時間排序的數據,利率、匯率和股價等都是時間序列數據。時間序列數據的時間間隔可以是分和秒(如高頻金融數據),也可以是日、周、月、季度、年以及甚至更大的時間單位。數據分析解決方案提供商 New Relic 在其博客上介紹了爲時間序列數據優化 K-均值聚類速度的方法。機器之心對本文進行了編譯介紹。
在 New Relic,我們每分鐘都會收集到 13.7 億個數據點。我們爲我們的客戶收集、分析和展示的很大一部分數據都是時間序列數據。爲了創建應用與其它實體(比如服務器和容器)之間的關係,以便打造 New Relic Radar 這樣的新型智能產品,我們正在不斷探索更快更有效的對時間序列數據分組的方法。鑑於我們所收集的數據的量是如此巨大,更快的聚類時間至關重要。
加速 k-均值聚類
k-均值聚類是一種流行的分組數據的方法。k-均值方法的基本原理涉及到確定每個數據點之間的距離並將它們分組成有意義的聚類。我們通常使用平面上的二維數據來演示這個過程。以超過二維的方式聚類當然是可行的,但可視化這種數據的過程會變得更爲複雜。比如,下圖給出了 k-均值聚類在兩個任意維度上經過幾次迭代的收斂情況:
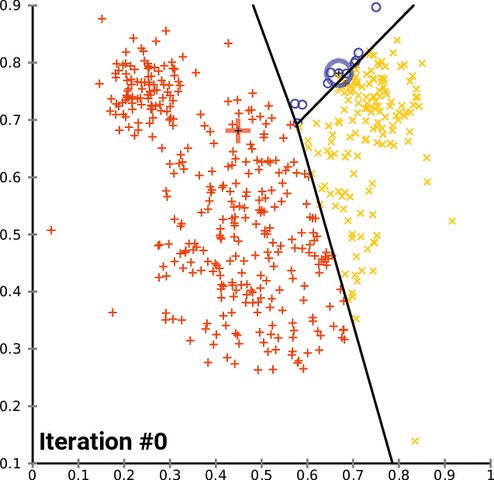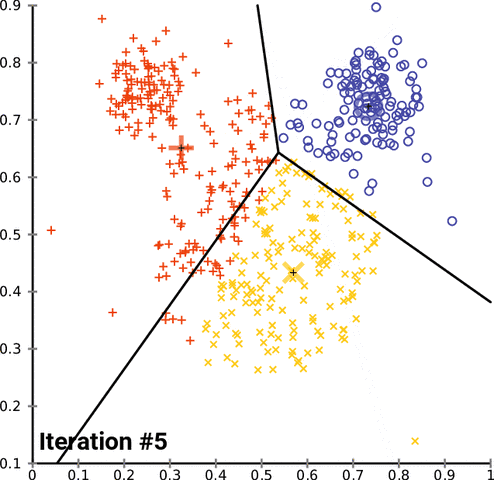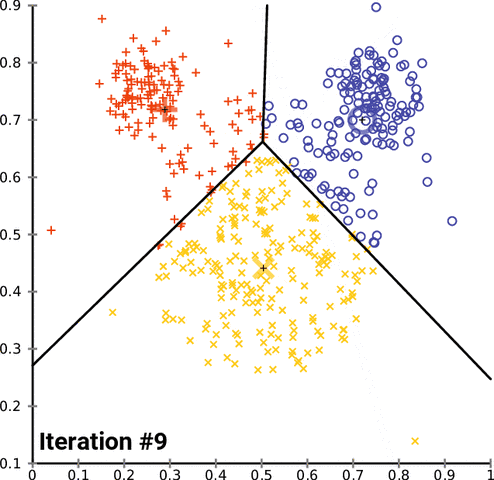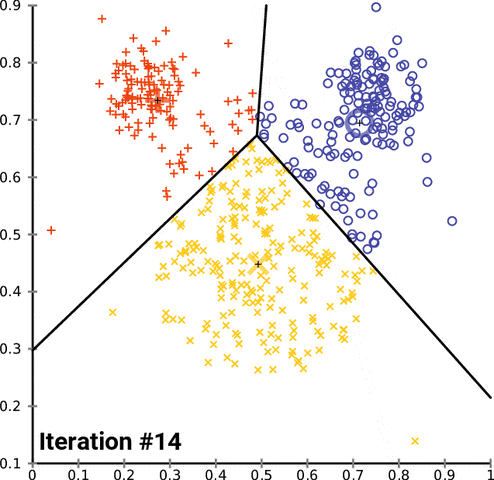
不幸的是,這種方法並不能很好地用於時間序列數據,因爲它們通常是隨時間變化的一維數據。但是,我們仍然可以使用一些不同的函數來計算兩個時間序列數據之間的距離因子(distance factor)。在這些案例中,我們可以使用均方誤差(MSE)來探索不同的 k-均值實現。在測試這些實現的過程中,我們注意到很多實現的表現水平都有嚴重的問題,但我們仍然可以演示加速 k-均值聚類的可能方法,在某些案例中甚至能實現一個數量級的速度提升。
這裏我們將使用 Python 的 NumPy 軟件包。如果你決定上手跟着練習,你可以直接將這些代碼複製和粘貼到 Jupyter Notebook 中。讓我們從導入軟件包開始吧,這是我們一直要用到的東西:
importtime
importnumpy asnp
importmatplotlib.pyplot asplt
%matplotlib inline
在接下來的測試中,我們首先生成 10000 個隨機時間序列數據,每個數據的樣本長度爲 500。然後我們向隨機長度的正弦波添加噪聲。儘管這一類數據對 k-均值聚類方法而言並不理想,但它足以完成未優化的實現。
n =10000
ts_len =500
phases =np.array(np.random.randint(0,50,[n,2]))
pure =np.sin([np.linspace(-np.pi *x[0],-np.pi *x[1],ts_len)forx inphases])
noise =np.array([np.random.normal(0,1,ts_len)forx inrange(n)])
signals =pure *noise
# Normalize everything between 0 and 1
signals +=np.abs(np.min(signals))
signals /=np.max(signals)
plt.plot(signals[0])
第一個實現
讓我們從最基本和最直接的實現開始吧。euclid_dist 可以爲距離函數實現一個簡單的 MSE 估計器,k_means 可以實現基本的 k-均值算法。我們從我們的初始數據集中選擇了 num_clust 隨機時間序列數據作爲質心(代表每個聚類的中心)。在 num_iter 次迭代的過程中,我們會持續不斷地移動質心,同時最小化這些質心與其它時間序列數據之間的距離。
defeuclid_dist(t1,t2):
returnnp.sqrt(((t1-t2)**2).sum())
defk_means(data,num_clust,num_iter):
centroids =signals[np.random.randint(0,signals.shape[0],num_clust)]
forn inrange(num_iter):
assignments={}
forind,i inenumerate(data):
min_dist =float('inf')
closest_clust =None
forc_ind,j inenumerate(centroids):
dist =euclid_dist(i,j)
ifdist <min_dist:
min_dist =dist
closest_clust =c_ind
ifclosest_clust inassignments:
assignments[closest_clust].append(ind)
else:
assignments[closest_clust]=[]
assignments[closest_clust].append(ind)
forkey inassignments:
clust_sum =0
fork inassignments[key]:
clust_sum =clust_sum +data[k]
centroids[key]=[m /len(assignments[key])form inclust_sum]
returncentroids
t1 =time.time()
centroids =k_means(signals,100,100)
t2 =time.time()
print("Took {} seconds".format(t2 -t1))
Took1138.8745470046997seconds
聚類這些數據用去了接近 20 分鐘。這不是很糟糕,但肯定算不上好。爲了在下一個實現中達到更快的速度,我們決定去掉儘可能多的 for 循環。
向量化的實現
使用 NumPy 的一大優勢是向量化運算。(如果你不太瞭解向量化運算,請參考這個鏈接:http://www.scipy-lectures.org/intro/numpy/operations.html)
k-均值算法要求每個質心和數據點都成對地進行比較。這意味着在我們之前的迭代中,我們要將 100 個質心和 10000 個時間序列數據分別進行比較,也就是每次迭代都要進行 100 萬次比較。請記住每次比較都涉及到兩個包含 500 個樣本的集合。因爲我們迭代了 100 次,那就是說我們總共比較了 1 億次——對於單個 CPU 而言算是相當大的工作量了。儘管 Python 是一種還算高效的語言,但效率還趕不上用 C 語言寫的指令。正是由於這個原因,NumPy 的大部分核心運算都是用 C 語言寫的,並且還進行了向量化以最小化由循環帶來的計算開銷。
我們來探索一下我們可以如何向量化我們的代碼,從而去掉儘可能多的循環。
首先,我們將代碼分成不同的功能模塊。這能讓我們更好地理解每個部分所負責的工作。接下來,我們修改 calc_centroids 步驟以便僅在質心上迭代(而不是在每個時間序列數據上)。這樣,我們將所有時間序列數據和一個質心傳遞給 euclid_dist。我們還可以預先分配 dist 矩陣,而不是將其當成一個詞典進行處理並隨時間擴展它。NumPy 的 argmin 可以一次性比較每個向量對。
在 move_centroids 中,我們使用向量運算去掉了另一個 for 循環,而且我們只在獨特的質心集上迭代。如果我們丟失了一個質心,我們就通過從我們的時間序列數據集中進行隨機選擇來加入合適的數字(這在實際應用的實踐中很罕見)。
最後,我們添加一個提前停止(early stopping)來檢查 k_means——如果質心不再更新,就停止迭代。
來看看代碼:
defeuclid_dist(t1,t2):
returnnp.sqrt(((t1-t2)**2).sum(axis =1))
defcalc_centroids(data,centroids):
dist =np.zeros([data.shape[0],centroids.shape[0]])
foridx,centroid inenumerate(centroids):
dist[:,idx]=euclid_dist(centroid,data)
returnnp.array(dist)
defclosest_centroids(data,centroids):
dist =calc_centroids(data,centroids)
returnnp.argmin(dist,axis =1)
defmove_centroids(data,closest,centroids):
k =centroids.shape[0]
new_centroids =np.array([data[closest ==c].mean(axis =0)forc innp.unique(closest)])
ifk -new_centroids.shape[0]>0:
print("adding {} centroid(s)".format(k -new_centroids.shape[0]))
additional_centroids =data[np.random.randint(0,data.shape[0],k -new_centroids.shape[0])]
new_centroids =np.append(new_centroids,additional_centroids,axis =0)
returnnew_centroids
defk_means(data,num_clust,num_iter):
centroids =signals[np.random.randint(0,signals.shape[0],num_clust)]
last_centroids =centroids
forn inrange(num_iter):
closest =closest_centroids(data,centroids)
centroids =move_centroids(data,closest,centroids)
ifnotnp.any(last_centroids !=centroids):
print("early finish!")
break
last_centroids =centroids
returncentroids
t1 =time.time()
centroids =k_means(signals,100,100)
t2 =time.time()
print("Took {} seconds".format(t2 -t1))
adding 1centroid(s)
early finish!
took 206.72993397712708seconds
耗時 3.5 分鐘多一點。很不錯!但我們還想完成得更快。
k-means++ 實現
我們的下一個實現使用了 k-means++ 算法。這個算法的目的是選擇更優的初始質心。讓我們看看這種優化方法有沒有用……
definit_centroids(data,num_clust):
centroids =np.zeros([num_clust,data.shape[1]])
centroids[0,:]=data[np.random.randint(0,data.shape[0],1)]
fori inrange(1,num_clust):
D2 =np.min([np.linalg.norm(data -c,axis =1)**2forc incentroids[0:i,:]],axis =0)
probs =D2/D2.sum()
cumprobs =probs.cumsum()
ind =np.where(cumprobs >=np.random.random())[0][0]
centroids[i,:]=np.expand_dims(data[ind],axis =0)
returncentroids
defk_means(data,num_clust,num_iter):
centroids =init_centroids(data,num_clust)
last_centroids =centroids
forn inrange(num_iter):
closest =closest_centroids(data,centroids)
centroids =move_centroids(data,closest,centroids)
ifnotnp.any(last_centroids !=centroids):
print("Early finish!")
break
last_centroids =centroids
returncentroids
t1 =time.time()
centroids =k_means(signals,100,100)
t2 =time.time()
print("Took {} seconds".format(t2 -t1))
early finish!
took 180.91435194015503seconds
相比於我們之前的迭代,加入 k-means++ 算法能得到稍微好一點的性能。但是,當我們將其並行化之後,這種優化方法才真正開始帶來顯著回報。
並行實現
到目前爲止,我們所有的實現都是單線程的,所以我們決定探索 k-means++ 算法的並行化部分。因爲我們在使用 Jupyter Notebook,所以我們選擇使用用於並行計算的 ipyparallel 來管理並行性(ipyparallel 地址:https://github.com/ipython/ipyparallel)。使用 ipyparallel,我們不必擔心整個服務器分叉,但我們需要解決一些特殊問題。比如說,我們必須指示我們的工作器節點加載 NumPy。
importipyparallel asipp
c =ipp.Client()
v =c[:]
v.use_cloudpickle()
withv.sync_imports():
importnumpy asnp
關於加載工作器更多詳情,請參閱 ipyparallel 上手指南:https://ipyparallel.readthedocs.io/en/latest/
在這一個實現中,我們的重點放在並行化的兩個方面。首先,calc_centroids 有一個在每個質心上迭代並將其與我們的時間序列數據進行比較的循環。我們使用了 map_sync 來將這些迭代中的每一個發送到我們的工作器。
接下來,我們並行化 k-means++ 質心搜索中一個相似的循環。注意其中對 v.push 的調用:因爲我們的 lambda 引用的數據,我們需要確保它在工作器節點上是可用的。我們通過調用 ipyparallel 的 push 方法來將該變量複製到工作器的全局範圍中,從而實現了這一目標。
看看代碼:
defcalc_centroids(data,centroids):
returnnp.array(v.map_sync(lambdax:np.sqrt(((x -data)**2).sum(axis =1)),centroids))
defclosest_centroids(points,centroids):
dist =calc_centroids(points,centroids)
returnnp.argmin(dist,axis=0)
definit_centroids(data,num_clust):
v.push(dict(data=data))
centroids =np.zeros([num_clust,data.shape[1]])
centroids[0,:]=data[np.random.randint(0,data.shape[0],1)]
fori inrange(1,num_clust):
D2 =np.min(v.map_sync(lambdac:np.linalg.norm(data -c,axis =1)**2,centroids[0:i,:]),axis =0)
probs =D2/D2.sum()
cumprobs =probs.cumsum()
ind =np.where(cumprobs >=np.random.random())[0][0]
centroids[i,:]=np.expand_dims(data[ind],axis =0)
returncentroids
t1 =time.time()
centroids =k_means(signals,100,100)
t2 =time.time()
print("Took {} seconds".format(t2 -t1))
adding 2centroid(s)
early finish!
took 143.49819207191467seconds
結果只有兩分鐘多一點,這是我們目前實現的最快速度!
接下來:更快!
在這些測試中,我們都只使用了中央處理器(CPU)。CPU 能提供方便的並行化,但我們認爲再多花點功夫,我們就可以使用圖形處理器(GPU)來實現聚類,且速度將得到一個數量級的提升。我們也許可以使用 TensorFlow 來實現,這是一個用於數值計算和機器學習的開源軟件。實際上,TensorFlow 已經包含了 k-均值實現,但我們基本上肯定還是需要對其進行調整才能將其用於時間序列聚類。不管怎樣,我們都不會停下尋找更快更高效的聚類算法的步伐,以幫助管理我們的用戶的數據。
原文鏈接:https://blog.newrelic.com/2017/10/23/optimizing-k-means-clustering/
本文爲機器之心編譯,轉載請聯繫本公衆號獲得授權。
責任編輯: